Commentaire d’article. Hepatitis B virus X protein identifies the Smc5/6 complex as a host restriction factor. Decorsières et al. Nature, 17 March 2016
Pendant près de 30 ans les chercheurs travaillant sur le virus de l’hépatite B (HBV) qui, rappelons-le, infecte de manière chronique environ 240 millions de personnes et est responsable de 50% des carcinomes hépatocellulaires (CHC) dans le monde1, se sont penchés sur le rôle de l’énigmatique protéine X/HBx, la bien nommée. En effet, si très rapidement il a été découvert que cette protéine pouvait aider à la transcription des ARN viraux à partir de l’ADNccc (c.à.d. ADN circulaire et covalemment clos qui est responsable de la persistance virale dans le noyau des cellules infectées)2,3, puis que la protéine pouvait être impliquée, par une myriade de mécanismes, dans la dérégulation de la physiologie cellulaire pouvant conduire à la transformation cellulaire3,4, ce n’est qu’aujourd’hui qu’une de ces fonctions régaliennes a été révélée dans un article paru dans l’édition du 17 mars 2016 de Nature5.
Pour rappel, HBx est une protéine qui ne ressemble à aucune autre, dont la structure cristalline n’est pas connue, et qui joue un rôle régulateur dans le cycle de vie du virus3. À ce jour, les certitudes que nous avions par rapport à sa/ses fonction(s) étaient qu’un virus délété du gène la codant (virus HBx-neg) ne peut pas se répliquer in vivo (modèle de la marmotte Américaine)6,7 et in vitro dans des hépatocytes non transformés (hépatocytes primaires humains et hépatocytes dérivés de progéniteurs hépatiques)8. Par ailleurs, nous savions qu’HBx n’était pas nécessaire à l’établissement du pool de cccDNA dans la cellule infectée, car un virus HBx-neg peut installer un réservoir d’ADNccc en cellules hépatocytaires, qui peut rester longtemps transcriptionnellement inactif et être « trans-complémenté » par un apport ectopique d’HBx8. Nous savions enfin qu’HBx n’était pas indispensable à la production de particules virales en cellules hépatocytaires transformées, car des stocks de virus HBx-neg peuvent être produits à très fort titre dans ces cellules9,10. Si HBx permet la transactivation de l’expression de gènes, sa spécificité d’action sur les gènes épisomiques viraux et/ou chromosomiques cellulaires est encore fortement débattue4,11,12. Les données suggérant qu’HBx peut être associé à des complexes transcriptionellement actifs et dépourvus d’enzymes/protéines permettant la répression génique ne sont quant à elles que corrélatives et pas vraiment fonctionnelles. Ainsi on ne savait pas jusqu’à ce jour pas si HBx était impliqué dans le recrutement actif de facteurs stimulant la transcription (ex: acétyltransférase, facteurs de transcription etc.…) ou s’il était impliqué dans l’élimination de facteurs négatifs (ex : histone-deacétylase, histone-méthyltransférase).
Jusqu’à présent un seul facteur cellulaire interagissant fortement et spécifiquement avec HBx a été identifié de manière convaincante et reproductible ; il s’agit de la protéine DDB1 (Damage DNA binding protein 1) qui est impliquée dans les processus de réparation de l’ADN et dans la dégradation de protéines cibles via son interaction avec le complexe CRL4 de la famille des « Cullin-RING ubiquitin ligase » (complexe CRL4 = CUL4 + Roc + E2 enzyme)13. En fait HBx se comporte comme une protéine DCAF (DDB1 Cullin Acessory Factor), c’est-à-dire une protéine accessoire faisant le lien entre la protéine cible à dégrader et le complexe DDB1-CRL4 et jouant ainsi un rôle crucial pour le choix de la cible13. Ainsi, il a été postulé depuis longtemp qu’HBx pourrait induire la dégradation d’au moins un facteur cellulaire restreignant la réplication d’HBV. Mais comment identifier un tel facteur qui est potentiellement rapidement dégradé parce qu’il interagit avec le complexe HBx-DDB1-CRL4 ?
Cette réponse nous a enfin été apportée par les auteurs de cet article5. En effet afin d’inhiber la dégradation du « facteur cellulaire restrictif » interagissant avec HBx, ils ont créé une protéine chimère HBx-DDB1 qui n’était plus capable d’interagir avec le complexe CRL4, et l’ont utilisé comme un appât pour définir l’interactome d’HBx. Avec cette approche, ils ont identifié le complexe Smc5/Smc6 comme étant le « facteur cellulaire restrictif » fixé par l’intermédiaire d’HBx et dégradé après ubiquitination via le complexe DDB1-CRL4. Le complexe Smc5/Smc6 est physiologiquement impliqué dans la régulation de la cohésion et la condensation des chromosomes pendant la mitose, ainsi que dans la recombinaison et la réparation de l’ADN14. Il est fortement exprimé dans les hépatocytes humains y compris pendant l’interphase (c.à.d. en dehors de la mitose).
Dans l’article, il est montré que ce complexe induit la répression transcriptomique sur des ADN de type épisomal, comme l’ADNccc d’HBV. Par des expériences de précipitation de la chromatine, les auteurs ont montré que le complexe Smc5/6 se fixe sur l’ADNccc en absence d’HBx. De plus, une perte de fonction par interférence ARN permet d’augmenter la transcription de l’ADNccc, alors qu’une surexpression de Smc5/6 au contraire renforce l’inhibition. Au final HBx permet donc la degradation de ce complexe et donc à l’ADNccc d’être transcrip-tionellement actif (voir Figure issue de la référence15). Ceci a été vérifié dans les modèles les plus pertinents pour étudier la réplication d’HBV, c.à.d. les hépatocytes primaires humains préparés à partir de résections de foie et dans le modèle des souris humanisées au niveau de leurs foies.
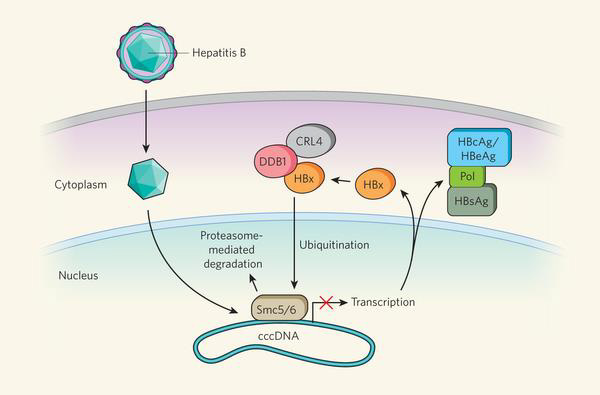
De nombreuses questions restent cependant en suspens. Comme le complexe Smc5/6 est composé de deux protéines Smc mais également de protéines non-Smc (Nse1 à Nse6)14, il n’a pas encore été déterminé quelle était exactement la protéine qui interagissait directement avec HBx. La connaissance de cette interface d’interaction sera importante afin d’identifier des molécules pouvant l’inhiber, et donc restaurer l’expression du complexe Smc5/6 et induire la répression transcritomique de l’ADNccc. Il semblerait que le complexe Smc5/6 soit un système de restriction de l’expression génique à partir de génomes épisomaux. HBV n’est pas le seul virus qui possède un génome épisomal. Il y a notamment les vecteurs de thérapie génique de type AAV (adenovirus asssociated virus) qui sont également maintenus sous cette forme. Or, nous savons que l’intensité et la durabilité de l’expression d’un « transgène médicament » peuvent être des éléments-clés de ce type de thérapie. Puisqu’il a été montré que la protéine HBx permet d’augmenter l’expression d’un transgène porté par un génome AAV16, il est probable que la restriction par Smc5/6 s’applique aussi aux vecteurs AAV. L’utilisation de vecteurs AAV pour délivrer un « transgène médicament » dans une cellule infectée par HBV semble donc opportune. Une autre question importante est relative au rôle physiologique de Smc5/6 ; en effet dans les cellules infectées, ce complexe est dégradé et ne peut plus remplir sa fonction s’agissant de la biologie des chromosomes14. Est-ce qu’il existe des redondances fonctionnelles, et est-ce que les complexes de type « cohésine » et « condensine », qui appartiennent à la même famille, prennent le relai ? Ou bien, est-ce que cette élimination permanente du complexe Smc5/6 peut avoir des conséquences pouvant conduire à terme à la transformation cellulaire ? Ceci devra être déterminé dans un futur proche.
- Busch, K. & Thimme, R. Natural history of chronic hepatitis B virus infection. Med Microbiol Immunol 204, 5-10, (2015).
- Belloni, L. et al. Nuclear HBx binds the HBV minichromosome and modifies the epigenetic regulation of cccDNA function. Proceedings of the National Academy of Sciences of the United States of America 106, 19975-19979, (2009).
- Seeger, C., Zoulim, F. & Mason, W. S. in Field’s Virology Vol. 2 (eds D.M. Knipe & P.M. Howley) Ch. 68, 2185 (Lippincott Williams & Wilkins, 2015).
- Guerrieri, F., Belloni, L., Pediconi, N. & Levrero, M. Molecular mechanisms of HBV-associated hepatocarcinogenesis. Seminars in liver disease 33, 147-156, (2013).
- Decorsiere, A. et al. Hepatitis B virus X protein identifies the Smc5/6 complex as a host restriction factor. Nature 531, 386-389, (2016).
- Zhang, Z., Torii, N., Hu, Z., Jacob, J. & Liang, T. J. X-deficient woodchuck hepatitis virus mutants behave like attenuated viruses and induce protective immunity in vivo. The Journal of clinical investigation 108, 1523-1531, (2001).
- Zoulim, F., Saputelli, J. & Seeger, C. Woodchuck hepatitis virus X protein is required for viral infection in vivo. Journal of virology 68, 2026-2030 (1994).
- Lucifora, J. et al. Hepatitis B virus X protein is essential to initiate and maintain virus replication after infection. Journal of hepatology 55, 996-1003, (2011).
- Ladner, S. K. et al. Inducible expression of human hepatitis B virus (HBV) in stably transfected hepatoblastoma cells: a novel system for screening potential inhibitors of HBV replication. Antimicrobial Agents and Chemotherapy 41, 1715-1720 (1997).
- Sells, M. A., Chen, M. L. & Acs, G. Production of hepatitis B virus particles in Hep G2 cells transfected with cloned hepatitis B virus DNA. Proceedings of the National Academy of Sciences of the United States of America 84, 1005-1009 (1987).
- Slagle, B. L. & Bouchard, M. J. Hepatitis B Virus X and Regulation of Viral Gene Expression. Cold Spring Harbor perspectives in medicine 6, (2016).
- van Breugel, P. C. et al. Hepatitis B virus X protein stimulates gene expression selectively from extrachromosomal DNA templates. Hepatology 56, 2116-2124, (2012).
- Minor, M. M. & Slagle, B. L. Hepatitis B virus HBx protein interactions with the ubiquitin proteasome system. Viruses 6, 4683-4702, (2014).
- Jeppsson, K., Kanno, T., Shirahige, K. & Sjogren, C. The maintenance of chromosome structure: positioning and functioning of SMC complexes. Nature reviews. Molecular cell biology 15, 601-614, (2014).
- Liang, T. J. Virology: The X-Files of hepatitis B. Nature 531, 313-314, (2016).
- Hosel, M. et al. Hepatitis B virus infection enhances susceptibility toward adeno-associated viral vector transduction in vitro and in vivo. Hepatology 59, 2110-2120, (2014).

