Commentaire d’article. Unique genomic profile of fibrolamellar hepatocellular carcinoma. Cornella et al, Gastroenterology 2015, 148 : 806-818
Jusqu’en 2014, il y a eu peu d’avancées dans la recherche sur le Carcinome Fibrolamellaire (CFL), depuis sa description en 1956 par Edmondson. Considéré comme une variante rare et intrigante de carcinome hépatocellulaire (CHC), sa pathogenèse est mal connue. Il se développe principalement chez l’enfant et l’adulte jeune sur foie non cirrhotique et ses caractéristiques morphologiques (travées de cellules oncocytaires séparées par de larges bandes fibreuses, fig.A) sont bien connues. Cependant, le CFL peut poser des problèmes de diagnostic différentiel avec la variante sclérosante du CHC, chez des sujets plus âgés, ou lorsque un CHC se développe sur un foie non fibreux. Curable uniquement par chirurgie et longtemps considéré comme de bon pronostic, la mise en évidence de récidives fréquentes et de son potentiel métastatique a conduit à effectuer des recherches génétiques et moléculaires nécessaires à la compréhension de la carcinogenèse du CFL, à l’identification de biomarqueurs et au développement d’alternatives thérapeutiques. L’avancée récente la plus marquante a été la découverte en 2014 d’un transcrit chimérique de fusion DNAJB1-PRKACA, très sensible (1) et très spécifique (2) du CFL, résultant d’une délétion de 400pb dans le chromosome 19, produisant une protéine de fusion qui maintient l’activité kinase de PRKACA.
Cet article s’inscrit dans cette dynamique récente sur le CFL. Il présente l’intérêt majeur (1) de réunir la plus grande cohorte multicentrique internationale de 78 CFL purs, revus indépendamment par trois pathologistes et parfaitement documentés sur le plan clinique (âge médian : 25 ans, 11- 65) et (2) d’avoir conduit des analyses génomiques complexes sur tissus fixés ou congelés, issus de cohortes d’entrainement et de validation, visant à mieux caractériser cette entité. Certains résultats sont originaux ; d’autres confirment ou complètent des données récentes.
Principaux résultats et commentaires :
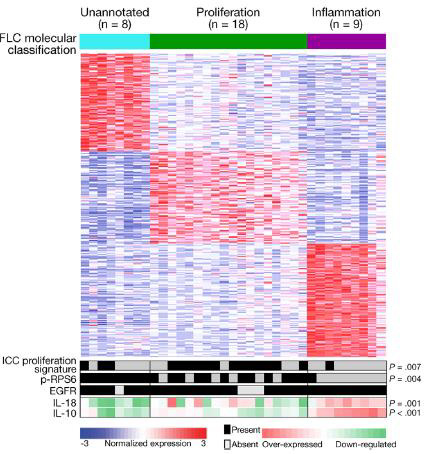
1 – identification de trois classes moléculaires, par une analyse globale de l’expression des gènes en mode non supervisé (Fig.B) :
- une classe proliférative majoritaire (41-51%), caractérisée par une surexpression de gènes décrits dans les CHC classés G2 (3) et dans les cholangiocarcinomes (CC) classés prolifératifs (4), impliqués dans la prolifération cellulaire et dans l’activation de la voie Akt-mTOR, voie dont la dérégulation déjà décrite dans le CFL (5) se traduit par l’expression immunohistochimique de p-RPS6
- une classe inflammatoire (26-41%), comportant une surexpression de gènes régulant l’inflammation hépatique, rapportés dans certains CC classés inflammatoires (4), et régulant la production de cytokines pro-inflammatoires, notamment IL-10 et IL-18
- une classe non décrite (unnanotated, 17-23%), dont la signature est constituée de gènes dont la dérégulation n’a pas été précédemment identifiée dans les cancers hépatiques (MAPKs, TGFB, PIK3, CTNNB1, RAS) mais a déjà été décrite dans le CFL (6).
De plus, une expression des marqueurs neuroendocrines (NTS, CALCA, PCSK1, DNER) et de la protéine EGFR est observée dans les 3 classes, corroborant des données récentes (7).
Cette répartition des CFL en 3 classes moléculaires est basée sur une signature génomique robuste. Son implication pronostique n’est pas encore démontrée mais une tendance à un meilleur pronostic est notée pour la classe non décrite.
2 – identification d’anomalies chromosomiques, moins nombreuses que dans les CHC et les CC, distribuées dans les trois classes, dans des loci souvent clés: amplifications focales impliquant des oncogènes (22%, dont 8q24.3 (RPL8), 6q27 (MLLT4) et 17q25.3) et délétions focales affectant des gènes suppresseurs de tumeurs (31%, dont 19p13.12, 19p13.3 (STK1), 22q13.32 et 11p15.5 (CDKN1C). La délétion focale 19p13.12 est responsable de la formation de la protéine de fusion DNAJB1-PRKACA. Dans cette étude, le transcrit chimérique de fusion est retrouvé dans 80% des CFL, confirmant son intérêt diagnostique et comme cible thérapeutique, bien que sa sensibilité ne soit pas de 100% comme initialement décrite.
3 – identification d’un profil mutationnel unique constitué de mutations somatiques peu ou pas décrites dans les cancers hépatiques, dont deux dans BRCA2, gène suppresseur de tumeur impliqué dans la réparation de l’ADN, présentes dans 4,2 % des CFL. Aucune des mutations couramment identifiées dans les CHC et les CC (TP53, CTNNB1, ARID1A, TTN, NFE2L2, AXIN1, EGFR, BRAF, KRAS, NRAS, IDH1, IDH2) n’est retrouvée dans le CFL.
4 – confirmation d’une différenciation mixte hépatocytaire et cholangiocytaire quasi universelle, avec un immunophénotype caractéristique HepPar1+ (98%) et CK7+ (90%), antérieurement rapporté (8). Les marqueurs des cellules progénitrices (Epcam, CK19) sont exprimés dans 38% des CFL (Fig.C), principalement dans la classe proliférative, posant la question d’une agressivité supérieure de cette classe. Ils sont absents dans la classe non décrite.
5 – identification d’une signature pronostique basée sur l’expression de 8 gènes relevants en matière de carcinogenèse (PEAR1, KRTAP, KLRD1, OSBPL8, RPL32, SLC26A11, RGS11, RAPGEF1) permettant d’établir un score prédictif de mortalité après résection appelé index de mortalité et de prédire le risque de récidive après résection. Aucun des patients avec une signature de mauvais pronostic ne se trouve dans la classe non décrite, suggérant une évolution plus favorable pour cette classe.

En conclusion : cette analyse génomique intégrée établit un profil du CFL, distinct des principaux cancers hépatiques, CHC et CC, et définit trois classes moléculaires dont la valeur pronostique reste à préciser. Elle confirme la valeur diagnostique du transcrit de fusion DNAJB1–PRKAC très sensible et hautement spécifique du CFL. Associées à la signature génomique pronostique, ces données pourraient permettre d’envisager des thérapies ciblées, définies sur la base des gènes candidats identifiés dans cette étude (DNAJB1–PRKAC, BRCA2, MLTT4, mTOR, EGFR).
Bibliographie
(1) Honeyman JN, Simon EP, Robine N, et al. Detection of a recurrent DNAJB1-PRKACA chimeric transcript in fibrolamellar hepatocellular carcinoma. Science 2014, 343:1010–1014.
(2) Graham RP, Jin L, Knutson DL, et al. DNAJB1-PRKACA is specific for fibrolamellar carcinoma. Mod Pathol 2015, 28:822–829.
(3) Boyault S, Rickman DS, de Reynies A, et al. Transcriptome classification of HCC is related to gene alterations and to new therapeutic targets. Hepatology 2007, 45:42–52.
(4) Sia D, Hoshida Y, Villanueva A, et al. Integrative molecular analysis of intrahepatic cholangiocarcinoma reveals 2 classes that have different outcomes. Gastroenterology 2013, 144:829–840.
(5) Riehle KJ, Yeh MM, Yu JJ, et al. mTORC1 and FGFR1 signaling in fibrolamellar hepatocellular carcinoma. Mod Pathol 2015, 28:103–110.
(6) Kannangai R, Vivekanandan P, Martinez-Murillo F, et al. Fibrolamellar carcinomas show overexpression of genes in the RAS, MAPK, PIK3, and xenobiotic degradation pathways. Hum Pathol 2007, 38:639–644.
(7) Malouf GG, Job S, Paradis V, et al. Transcriptional profiling of pure fibrolamellar hepatocellular carcinoma reveals an endocrine signature. Hepatology 2014, 59:2228–2237.
(8) Ward SC, Huang J, Tickoo SK, et al. Fibrolamellar carcinoma of the liver exhibits immunohistochemical evidence of both hepatocyte and bile duct differentiation. Mod Pathol 2010, 23:1180–1190.

